發布時間:2025-11-07
2025年10月22日,Genome Research在線發表中國科學院廣州生物醫藥與健康研究院細胞命運研究組的研究論文,題為“Polyomino reconstructs spatial transcriptomic profiles with single-cell resolution via region-allocation method”,該論文被選為Genome Research第11期的封面文章。研究提出了一種全新的空間數據整合算法 Polyomino,能夠在單細胞分辨率下高效、精準地重建空間轉錄組圖譜。
在生命體中,細胞并非孤立存在,它們需要根據空間位置相互通信,形成精密的組織與器官。空間轉錄組學作為揭示這一細胞藍圖的關鍵技術,近年來發展迅速。然而,現有的單細胞數據與空間數據整合方法在面對動輒百萬級規模的細胞時,往往受限于運算效率和噪聲干擾,難以兼顧速度與精度。
研究團隊借鑒圖像處理中“感興趣區域”(ROI)的概念,提出通過多層次區域約束來分配細胞位置。與傳統方法直接分配不同,Polyomino先將空間數據劃分為多個格子,利用水平/垂直條帶、空間聚類等多層區域化信息約束細胞定位,再在格子內部進行精細匹配。這一設計不僅顯著降低了計算量,還能有效抵御常見的細胞分割誤差與細胞類型比例失衡等噪聲干擾。
在一系列模擬和真實數據的評估中,Polyomino表現出色:
a)?計算效率高:在整合約1.5萬單細胞和近2萬空間點的數據時,僅用141秒完成,比現有算法快10到1000倍,是目前唯一能夠在單次運行中處理百萬級細胞整合的方法。
b)?精度與魯棒性兼備:在多種噪聲場景(如分割誤差、比例偏差)下保持高精度;在小鼠胚胎和大腦皮層的驗證中,Polyomino均能準確重建細胞的空間分布。
c)?應用廣泛:在結直腸癌肝轉移組織的分析中,Polyomino揭示了cDC(常規樹突狀細胞)在腫瘤與旁腫瘤區的空間亞群差異,并識別出與血管生成相關的免疫信號網絡。這為進一步理解腫瘤微環境提供了新視角。
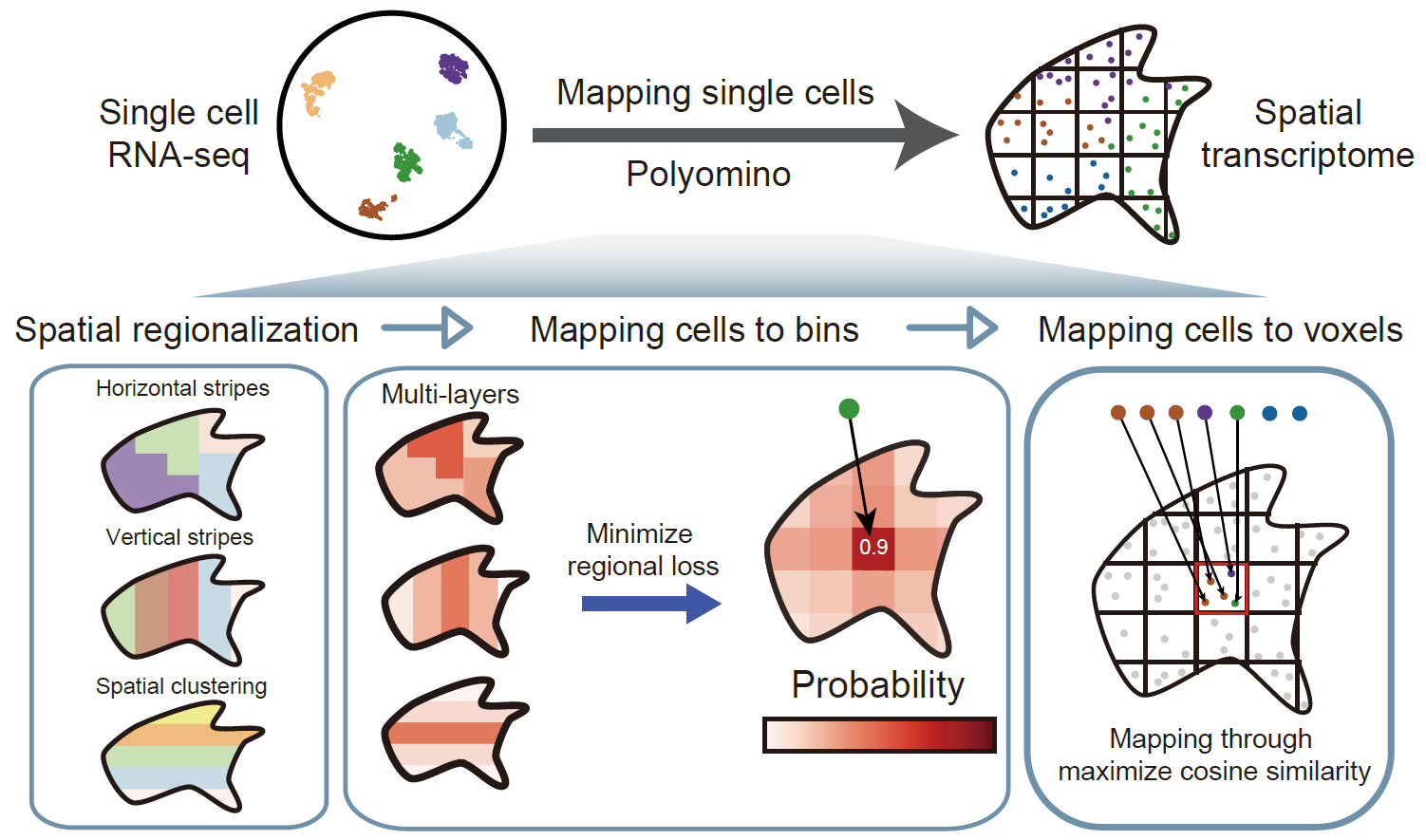
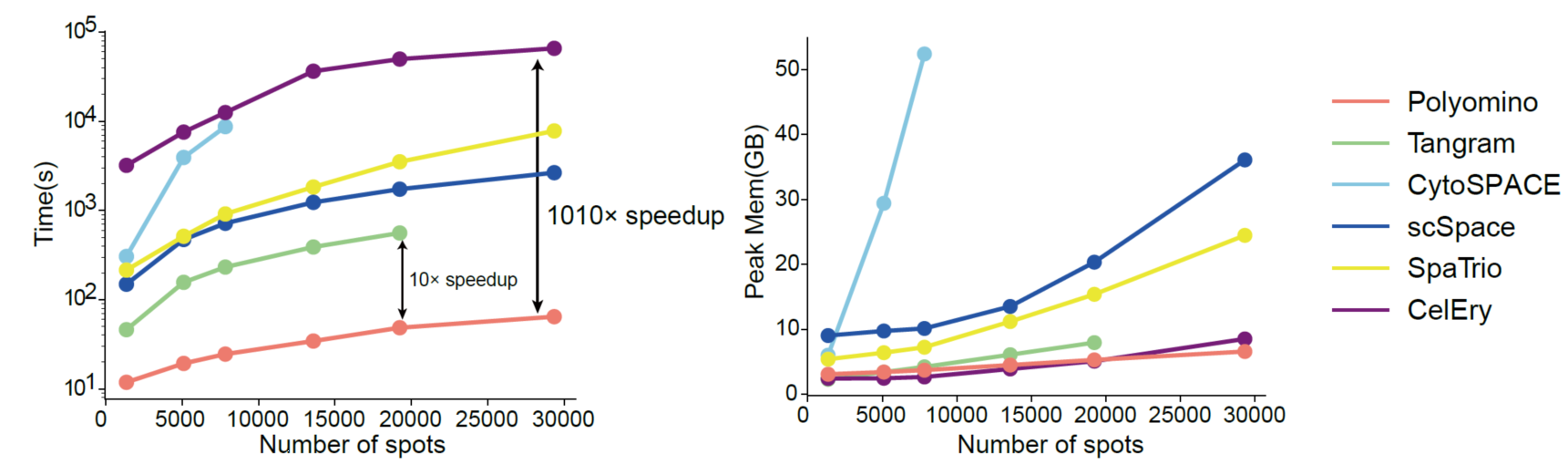
Polyomino通過多層區域約束實現單細胞空間定位,顯著提高了計算效率
綜上,Polyomino不僅突破了大規模數據整合的計算瓶頸,還為研究組織發育、疾病機制乃至未來臨床應用提供了更高分辨率的工具。研究者認為,隨著單細胞與空間組學數據量的持續增長,Polyomino有望成為構建全景細胞圖譜、解析器官發生與病理變化的重要方法學支撐。
中國科學院廣州生物醫藥與健康研究院博士蔡佺佑、中國科學院廣州生物醫藥與健康研究院副研究員林立惠與廣東第二師范學院劉欣博士為該論文共同第一作者。中國科學院廣州生物醫藥與健康研究院研究員陳捷凱、中國科學院廣州生物醫藥與健康研究院副研究員林立惠為該論文的共同通訊作者。該研究工作是人類細胞譜系大科學研究設施系統四的建設內容之一,也得到了國家重點研發計劃、國家自然科學基金、廣州實驗室重大專項、廣東省科技計劃項目及香港特區政府Health@InnoHK計劃等支持。

附件下載:

